
除了字典中包含的由 注釋的首字母縮寫詞之外,還可以在每個文檔的基礎上檢測到新穎的首字母縮寫詞聲明。當檢測到首字母縮寫詞定義(形式為“物種(首字母縮略詞)”時,其中物種在字典中,首字母縮寫詞是大寫字母、數字或連字符的序列),該首字母縮寫詞的所有后續出現也會在文檔中標記。
刪除常用英語單詞
基于一個簡單的物種名稱列表,這些物種名稱在不提及物種時通常出現在英語中(參見附加文件3),我們刪除了列表中包含物種術語組合的任何提及 . 這消除了“spot”(對于 )和“permit”(對于 )等同義詞,并大大減少了系統產生的誤報數量。
為模棱兩可的提及分配概率
最后,任何仍然模棱兩可的提及都被分配了提及特定物種的可能性的概率。模糊提及的概率基于所有 MEDLINE 和 PubMed Central 全文文檔的開放訪問子集中所涉及物種的明確提及的相對頻率。首字母縮寫詞的概率基于 檢測到的首字母縮寫詞定義的相對頻率(見上文)。例如,對于模棱兩可的提及“C. elegans”,出現秀麗隱桿線蟲的概率會非常高,而出現Crella elegans的概率會很高會低很多。對于首字母縮略詞“HIV”(可能同時指“人類免疫缺陷病毒”,更不常見的是“希波克拉底無關變量”),它指代“人類免疫缺陷病毒”的可能性非常高。
這些概率啟用了另一種啟發式消歧形式:在模棱兩可的提及具有高于給定截止值(例如 99%)的概率的物種替代的情況下,提及可以完全消除該物種的歧義(例如術語“C. elegans”可以被消除為 elegans)。同樣,如果所有與物種相關的提及概率之和小于給定閾值(例如 1%),則可以刪除提及;這可能發生在首字母縮略詞中,在 99% 以上的情況下,首字母縮略詞用于非物種術語。這些級別在準確性和模糊性最小化之間進行了權衡,并且可以在標記后根據用戶的個人需求進行調整。
輸入和輸出格式
能夠處理各種文檔 XML 格式,包括 MEDLINE XML、PMC XML、Biomed Central XML和 Open Text Mining XML。此外,它還可以處理來自本地存儲文件和遠程數據庫服務器的純文本文檔。物種名稱識別結果可以存儲到基于對峙的制表符分隔值文件、XML 文檔、HTML 文檔(用于結果的簡單可視化)和遠程 MySQL 數據庫表中。
用于物種標記的文檔集
在整個工作中,使用了三個不同的文檔集來識別和規范物種名稱。對于所有集合,2008 年之后發布的任何文檔都被刪除,以創建固定和可重復的文檔集合,并避免在項目過程中因數據庫記錄更新而可能出現的差異。
醫療線
MEDLINE 是 PubMed 文章摘要的主要數據庫,包含超過 1800 萬條條目。然而,許多條目實際上并不包含任何摘要。如果僅計算截至 2008 年底發表的包含摘要的條目,則文件數量剛剛超過 990 萬份。
PubMed Central 開放獲取子集
PMC 免費提供超過一百萬篇全文文章。不幸的是,其中只有大約 10%(截至 2008 年底發布了 105,106 篇)是真正的開放訪問并可用于不受限制的文本挖掘。此 PMC 的開放存取 (OA) 子集中的文章在此稱為“PMC OA”。PMC OA 中的大部分文章都是基于 XML 文件,但有些是通過掃描非數字文章(29,036 個文檔)的光學字符識別(OCR)創建的,還有一些是通過轉換便攜式文檔格式(PDF ) 文檔到文本(9,287 個文檔)。我們注意到,對于使用 OCR 或 pdf 到文本軟件生成的 PMC OA 文檔,不會從這些文檔中刪除參考。正因為如此,出現在參考標題中的物種名稱可能會被標記。對于所有其他文件(MEDLINE、即不處理參考標題)。
PMC OA 的摘要
PMC OA 集中所有文章的摘要形成一個稱為“PMC OA abs”的集。PMC OA 摘要是從 PMC OA XML 文件的摘要部分獲得的,或者如果 XML 文件中不存在這樣的部分,則從相應的 MEDLINE 條目獲得(當文章是通過 OCR 或 pdf 到文本工具生成時會發生這種情況) . PMC OA 摘要包含 88,962 篇文檔,明顯少于 PMC OA 中的文檔數量(105,106 篇)。這是因為并非所有 PMC 文章都被 MEDLINE 索引,因此一些 OCR 或 pdf 轉文本文檔沒有對應的 MEDLINE 條目,使得準確提取摘要不可行。在 88,962 篇摘要中,有 65,739 篇(74%)是從 XML 文檔中提取的,其余部分是從相應的 MEDLINE 文檔中提取的。
PMC OA 全文文檔集的劃分
如上一節所述,不可能可靠地提取 PubMed Central 中大約五分之一的全文文章的摘要,因為它們在 PMC XML 或相應的 MEDLINE 條目中沒有摘要部分。我們選擇不從我們的分析中刪除這些全文文章,因為它們包含 PubMed Central 中的大量文檔子集,并且它們的排除可能會使我們的結果產生偏差。但是,它們的包含使得基于 PMC OA 摘要和所有 PMC OA 全文文檔的結果的直接比較變得困難,因為 PMC OA 全文集中存在一些文檔,而 PMC OA 摘要集中缺少這些文檔。為了在文檔層面解決這個問題,我們創建了“PMC OA full (abs)”集,其中包含可以提取摘要的 88,962 個全文文檔,允許直接比較完全相同文章的全文文檔和摘要。不幸的是,該文檔集仍然不允許在摘要和全文之間進行直接提及級別的比較,因為來自 MEDLINE 條目的偏移坐標和 PMC OA 全文文檔不兼容。因此,我們創建了“PMC OA full (xml)”集,該集僅包含 65,739 個全文文檔,其中可以從相應的 PMC XML 文件中提取摘要。使用此 PMC OA 全文 XML 集,還可以在相同偏移坐標上對相同文檔集執行提及級別比較。我們注意到“PMC OA”是指完整的 105,106 個全文文檔集,我們也可以將其表示為“PMC OA full (all)”。
用于評估的文檔集
目前,不存在專門針對物種提及進行注釋的生物醫學文檔的開放訪問語料庫。因此,我們創建了許多自動生成的評估集,以分析 和其他物種名稱標記軟件的準確性。由于它們所基于的數據的性質,許多這些評估集只能在文檔級別進行分析。此外,這些自動生成的評估集都不是基于專門為注釋物種提及而創建的數據。正因為如此,我們創建了一個為物種提及手動注釋的全文文章的評估集。每個評估集覆蓋的文檔、物種和標簽的數量如表1所示完整的手動注釋文檔可以在項目網頁上找到。
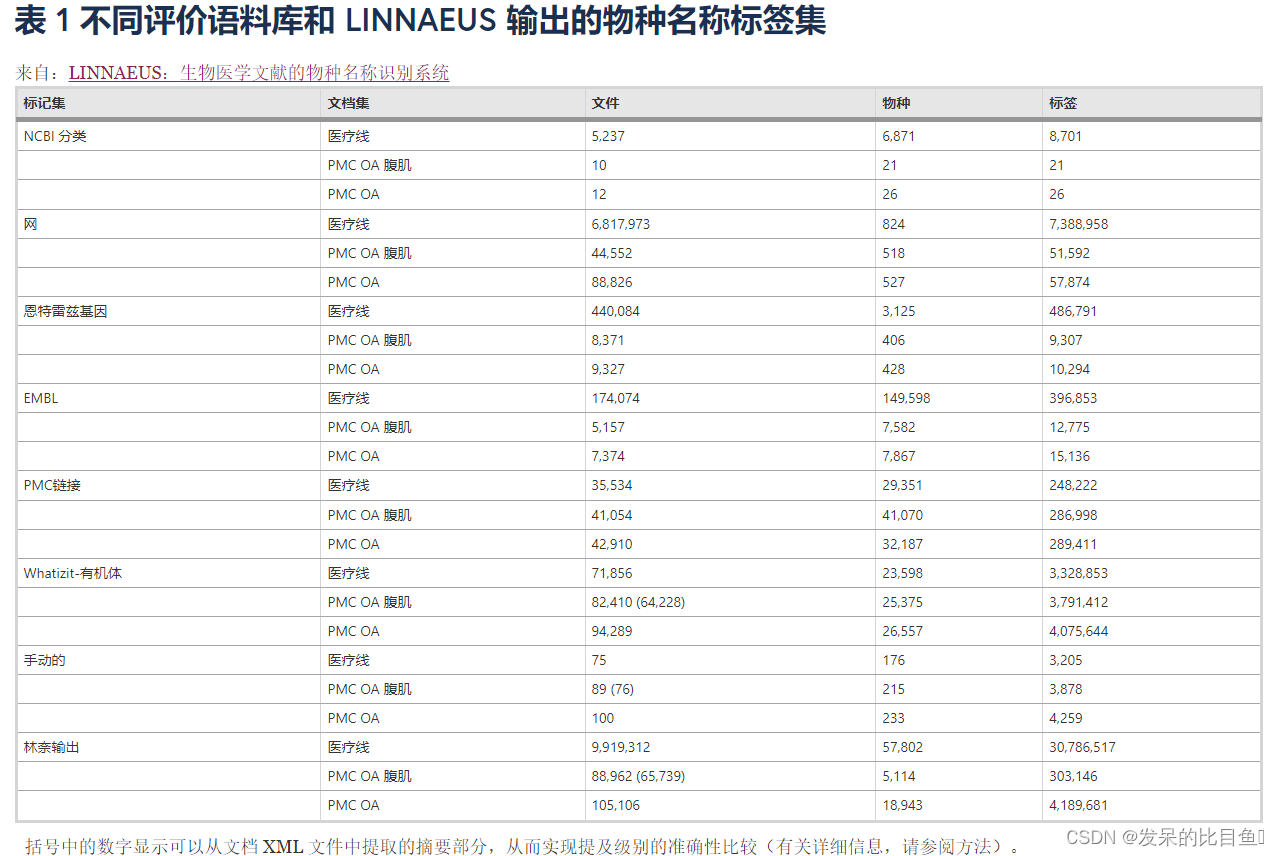
NCBI 分類引文

NCBI 分類中的一些物種條目包含對討論該物種的研究文章的引用。對于這些文件,我們假設該物種最有可能在文章的某處被提及,從而使相對回憶成為一種有用的衡量標準。NCBI 分類引文于 2009 年 6 月 1 日下載。
醫學主題詞條
MEDLINE 中的每篇文章都有相關的 MeSH 術語,指定文章中討論的主題。這些術語的一個子集與物種有關,并且可以通過統一醫學語言系統 (UMLS) 映射到 NCBI 分類物種條目。然而,由 MeSH 術語表示的物種數量是有限的。總共只有 1,283 個物種的 MeSH 術語,在 MEDLINE 的 MeSH 標簽中實際出現的物種只有 824 個。此外,賦予文章的 MeSH 術語并不能保證該術語在文檔中明確提及。此外,預計文檔中提及的總物種中只有一小部分會在 MeSH 標簽中表示(只有所謂的焦點物種),導致使用該語料庫的精度估計不如召回信息量大。
Entrez 基因條目
MEDLINE 中的每篇文章都有相關的 MeSH 術語,指定文章中討論的主題。這些術語的一個子集與物種有關,并且可以通過統一醫學語言系統 (UMLS) 映射到 NCBI 分類物種條目。然而,由 MeSH 術語表示的物種數量是有限的。總共只有 1,283 個物種的 MeSH 術語,在 MEDLINE 的 MeSH 標簽中實際出現的物種只有 824 個。此外,賦予文章的 MeSH 術語并不能保證該術語在文檔中明確提及。此外,預計文檔中提及的總物種中只有一小部分會在 MeSH 標簽中表示(只有所謂的焦點物種),導致使用該語料庫的精度估計不如召回信息量大。
EMBL 記錄
與 Entrez 基因記錄類似,許多 EMBL序列記錄還包含有關該序列來自哪個物種以及該序列是在哪篇文章中報道的信息。假設在報告核苷酸序列的論文中明確提到了物種,這可以提取物種-文章映射。然而,與 Entrez 基因集一樣,這并不能保證,除了具有報告序列的物種之外,討論的任何物種都不會出現在評估集中(再次導致精確測量無信息)。該評估集使用了 EMBL 的 r98 版本。
PubMed 中央鏈接
盡管沒有在任何出版物中描述,NCBI 對 PMC 中包含的全文文章進行物種識別文本挖掘。這些分類“鏈接”可以在查看 PMC 上的文章時訪問,也可以通過 NCBI e-utils Web 服務下載。通過下載這些鏈接,可以創建與召回率和精度相關的評估集(盡管僅在文檔級別)。PMC 鏈接數據于 2009 年 6 月 1 日下載。
為了評估提及級別的準確性并將 與另一個物種名稱識別系統進行基準比較,PMC OA 集中的所有文檔都通過 Web 服務管道發送。不幸的是, Web 服務無法處理大約 10% 的 PMC OA 文檔(參見表1),因此無法進行比較。 標記于 2009 年 6 月 25 日執行。
人工標注的金標準語料庫
由于所有前面描述的評估集都受到它們沒有專門為物種名稱注釋的事實的限制,因此很明顯需要這樣一個集來測量 的真實準確性。因為沒有這樣的評估集可用,所以從 PMC OA 文檔集中隨機選擇了 100 個全文文檔并為物種提及進行了注釋。由于這項工作的重點是物種而不是屬或其他更高階的分類單位,因此語料庫僅針對物種進行了注釋(除了在提及物種時錯誤地使用了屬名的情況)。
所有提及的物種術語均手動注釋并標準化為預期物種的 NCBI 分類 ID,但作者未提及該物種的術語除外。一個常見的例子是“Fisher 精確檢驗”(“Fisher”是Martes 的同義詞,但在這種情況下指的是發明統計檢驗的 Ronald Aylmer Fisher 爵士)。在 NCBI 分類中不存在物種 ID 的情況下(主要發生在特定物種菌株中),它們的物種 ID 為 0(在 NCBI 分類中不使用)。
帶注釋的提及也被分配到以下類別,這些類別表明提及的特定特征,可用于評估分析:
(一)詞匯類別:
提及可能屬于多個類別(例如,它可能既用作修飾符又可能拼寫錯誤),或者根本不屬于任何類別(即只是普通提及,這是最常見的情況)。表2顯示了與每個類別相關的物種標簽數量的摘要。這些類別可以深入了解物種名稱在文獻中拼寫錯誤或使用不正確的頻率。它們還可以對 或針對該語料庫評估的任何其他軟件所做的任何預測錯誤進行更深入的分析。在該語料庫中注釋的 4259 個物種中,72% (3065) 是常用名稱,這加強了在處理生物醫學研究文章時能夠準確識別常用名稱的重要性。
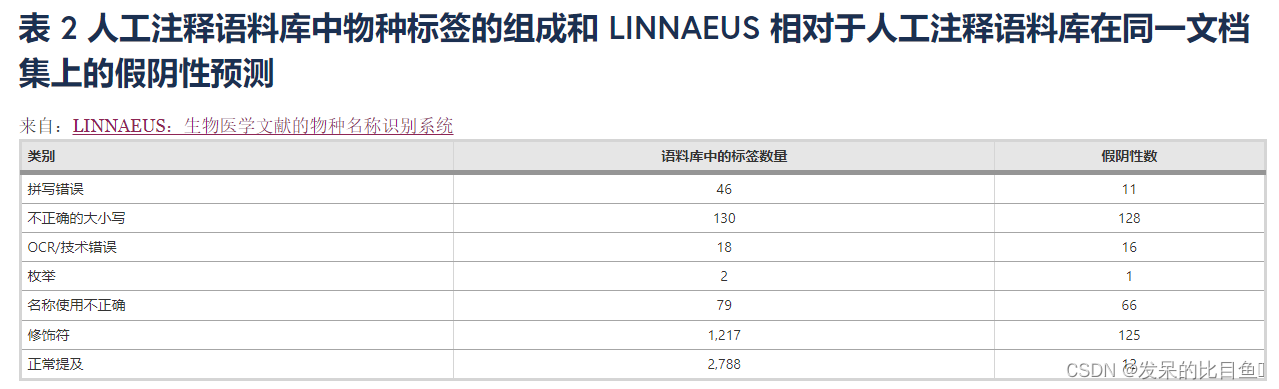
為了估計手動注釋的可靠性,10% 的語料庫(10 個文檔)也由第二個注釋器注釋,并計算了注釋器間協議 (IAA)。總共有 406 個物種提及在 10 個文件中由至少一個注釋者注釋。在這 406 次提及中,368 次被兩個注釋器(提及位置和物種標識符)相同地注釋。Cohen 對注釋者間一致性的 k 度量 [ 53 ] 計算為 k = 0.89。IAA 分析的詳細信息可在附加文件4中找到。
績效評估
將 生產的標簽與評估參考集中的標簽進行比較,以確定系統的性能。如果特定標簽同時出現在 集和參考集中,則稱為真陽性(TP);如果它僅出現在 集中,則稱為誤報 (FP);如果它僅出現在參考集中,則稱為假陰性(FN)。這在文檔級別(不考慮文檔中標簽的位置)和提及級別(標簽位置必須完全匹配)上執行。對于信息僅在文檔級別可用的評估集,不執行提及級別評估。在不明確提及的情況下,如果提及至少包含“真實”物種,則該提及被視為 TP(并且,對于提及水平分析,位置正確)。我們注意到 試圖識別文件中提到的所有物種,因此報告的物種數量沒有限制。
結果
我們將 系統應用于 2008 年或之前發表的近 1000 萬篇 MEDLINE 摘要和超過 100,000 篇 PMC OA 文章(表1)。使用四個 Intel Xeon 3 GHz CPU 內核和 4 GB 內存,MEDLINE 的文檔集標記大約需要 5 小時,PMC OA 摘要需要 2.5 小時,PMC OA 需要 4 小時。(我們注意到影響處理時間的主要因素是 Java XML 文檔解析而不是實際的物種名稱標記。)這些物種標記實驗遠遠超過了任何先前報告的規模,并代表了文本挖掘在整個 PMC OA 語料庫中的第一個應用。在 MEDLINE 中檢測到超過 57,000 個不同物種的超過 3000 萬個物種標簽,在 PMC OA 中檢測到近 19,000 個物種的超過 400 萬個物種標簽。 在 74% 的 MEDLINE 文章、72% 的 PMC OA 摘要和 96% 的 PMC OA 全文文章中識別出物種。從NCBI分類詞典中的物種總數來看,15%的NCBI詞典中的物種被在MEDLINE中找到,1.3%在PMC OA摘要中找到,4.9%在PMC OA全文中找到文章。MEDLINE 或 PMC OA 摘要中的物種名稱密度分別比 PMC OA 全文文章低 30 倍和 3 倍;相對于全文文檔,兩組摘要中物種提及的密度都低 11 倍。
MEDLINE 和 PubMed Central 中提到的物種的歧義
在所有 MEDLINE 和 PMC OA 中,11-14% 的物種提及是模棱兩可的。因此,物種名稱歧義的水平與基因名稱中的跨物種歧義處于相同的順序,并表明某種形式的消歧對于準確的物種名稱規范化是必要的。表3顯示了 消歧步驟之前和之后的標記文檔集的歧義級別. 歧義級別的計算方法是歧義提及的數量除以提及的總數,其中當提及映射到多個物種時,會計算歧義提及。消歧方法“無”顯示任何消歧之前的值;“earlier”通過掃描文檔中較早的明確提及來消除歧義,為了比較,“whole”通過掃描整個文檔中的明確提及來消除歧義。“嚴格”消歧不考慮正確物種提及的相關概率,而“近似”表示對單個物種具有高于 99% 概率或所有物種概率之和低于 1% 的任何提及的消歧。
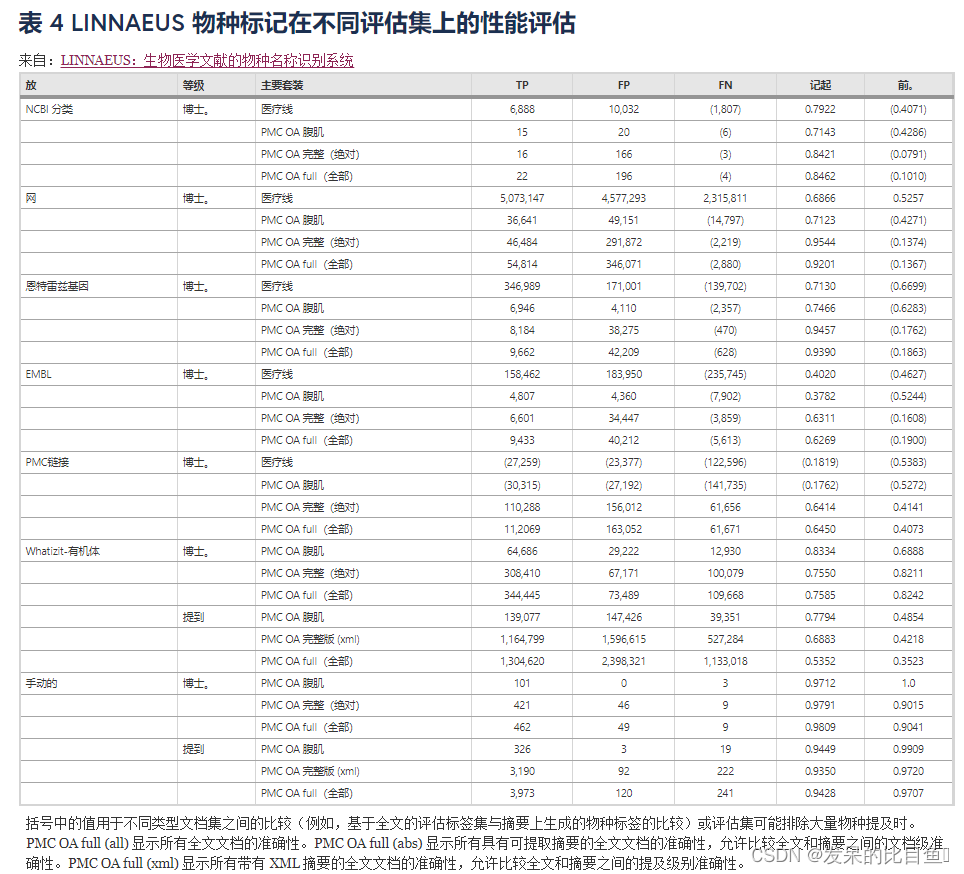
評估 物種名稱標記
與評估集中的物種相比, 發現的物種提及的評估如表4所示. 對于文檔級評估集(NCBI 分類參考、MeSH 標簽、Entrez 基因參考、EMBL 參考和 PMC 鏈接),文檔級標簽直接與 在 MEDLINE、PMC OA 摘要或 PMC OA 中找到的標簽進行比較文件。對于提及級評估集( 輸出和手動注釋集),僅在評估集和 PMC OA XML 之間直接比較標簽,因為 PMC OA XML 是唯一與評估集在相同偏移坐標上的文檔集(見方法)。對于自動生成的集合,我們在評估集中如何注釋物種的背景下解釋召回和精度,以提供對假陽性和假陰性的定性分析。對于人工標注的金標準評估集,
討論
物種名稱識別和規范化越來越被認為是文本挖掘和生物信息學中的一個重要主題,不僅因為它可以為最終用戶提供直接優勢,而且還可以指導其他軟件系統。雖然之前已經報道了許多執行物種名稱識別和/或科學名稱和同義詞標準化的工具,這里介紹的工作以多種獨特的方式為該領域做出了貢獻。其中包括強大的、開源的、獨立的應用程序的可用性(其他工具要么不公開提供,只能作為 Web 服務提供,要么不能識別常用名稱)、物種標記的規模(所有 MEDLINE 和 PMC OA 直到2008)、評估的深度和嚴謹性(其他工具不針對規范化的數據庫標識符進行評估,或者僅限于少量文檔樣本)和準確性(與其他可用工具相比, 表現出更好的性能,主要是由于更好地處理含糊不清的提及和包含其他同義詞)。此外,我們提供第一個開放訪問,
評估物種名稱識別軟件需要人工注釋的金標準
任何生物信息學應用程序的相對性能僅與與之比較的評估集一樣好。在物種名稱識別軟件的情況下,在當前工作之前,沒有開放訪問的生物醫學文本中物種名稱注釋的手動注釋數據集作為評估的黃金標準。在這個項目中,我們研究了四種不同的自動生成的評估集(NCBI 分類引文、MeSH 標簽、Entrez 基因參考、EMBL 引文),這些評估集基于策展的文檔-物種對。我們還根據使用文本挖掘軟件(PMC 和 )預測的文檔物種對研究了兩個不同的自動生成的評估集。盡管當文檔集和評估集屬于同一類型時,可以解釋 的召回(例如全文),由于在任何這些評估集中對物種提及的不完整或不完善的注釋,我們的系統的精度無法準確評估。我們得出結論,從“次要”來源(例如文檔基因(例如Entrez 基因)或文檔序列(例如EMBL)映射)自動推斷出的文檔-物種映射評估集在評估物種名稱識別軟件中的價值有限。
由于自動生成的評估集的固有局限性(包括物種名稱的不完整注釋或不正確的消歧),因此創建了手動注釋的評估語料庫。對手動注釋評估語料庫的評估顯示, 的性能非常好,在提及級別上具有 94.3% 的召回率和 97.1% 的準確率,在文檔級別上具有 98.1% 的召回率和 90.4% 的準確率。沒有一個自動生成的評估集能接近揭示使用 進行物種名稱識別的這種精度水平。這些結果強調了我們手動注釋的黃金標準評估集的重要性,并建議在自動生成的評估集上評估其他系統可能低估了系統精度。擁有高質量評估集的一個有趣觀察是,召回率高于文檔級別的準確率,而準確率高于提及級別的召回率。造成這種情況的一個原因是,當作者使用非標準或拼寫錯誤的名稱時,他們通常會在整個文檔中多次使用這些名稱,導致在提及級別上出現多個誤報,但僅在文檔級別上出現一次。相反,誤報在文檔中更分散,導致提及和文檔級別評估的誤報計數差異很小。
提高全文文章中物種名稱識別的準確性
目前絕大多數文本挖掘研究都是針對生物醫學文章的摘要進行的,因為它們在 PubMed 中免費提供,分析所需的計算資源較少,并且被認為包含最高密度的信息。然而,越來越多的證據表明,全文文章的信息檢索效果更好,因為生物醫學術語的覆蓋率高于摘要。我們的物種名稱識別結果支持這一結論,對于大多數測試的評估集,全文文章的物種名稱召回率高于摘要(表4) 并且幾乎所有 (96%) 全文文章都被標記為至少一個物種名稱。對全文文章進行術語識別的好處在物種名稱的情況下可能特別有用,因為與疾病、基因或化學品和藥物的術語相比,生物術語在生物醫學文檔的不同部分中的分布似乎更加統一。
我們的結果還清楚地表明,通過搜索明確提及來消除物種提及的歧義在全文文章中比在摘要中更成功。因此,正如之前發現的基因名稱,全文覆蓋率的增加對物種名稱消歧有額外的好處,因為在處理全文文章時,消歧算法可以獲得更多信息。有趣的是,我們發現無論是在文本的前面還是在整個文本中掃描明確提及,歧義的程度都會下降,這可能是因為文章的材料和方法部分通常位于論文的末尾。在搜索明確提及后,我們發現生物醫學文本中物種名稱的歧義水平很低(3-5%),如果可以容忍少量錯誤,可以使用概率方法進一步降低(1-3%)。
結論
我們開發并評估了一個強大的開源軟件系統 ,它可以快速準確地識別生物醫學文件中的物種名稱,并將它們規范化為 NCBI 分類中的標識符。 系統的低歧義性、高召回率和高精度使其非常適合生物醫學文本中的自動物種名稱識別。生物醫學領域的 物種識別可以通過包含細胞系名稱來增強 [ 67 ],這些名稱通常充當產生它們的物種的生物代理。 也可能在其他問題領域表現良好,例如生態學和分類學文獻,前提是提供高質量的物種名稱詞典(例如 [ 68]),盡管這仍然是未來研究的開放領域。進一步開發 以在生物醫學文獻之外更廣泛地應用可能需要與其他方法集成,例如基于規則的物種名稱識別系統(例如 ),我們目前的目標是在未來提供此類方法的實現,以便能夠使用 提供的文件處理方法。 的可用性現在為在文本中使用物種名稱的下游應用程序提供了機會,包括將物種名稱集成到更大的生物信息學管道中,生物醫學文本中物種名稱的語義標記,以及跨物種名稱使用趨勢的數據挖掘文件和時間。
*請認真填寫需求信息,我們會在24小時內與您取得聯系。